检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
2016年12月29日,william hill中文网伊成器研究组应邀在 《Nature Methods》 杂志发表题为“Epitranscriptome sequencing technologies: decoding RNA modifications” 的综述论文(doi:10.1038/nmeth.4110)。
表观转录组学(epitranscriptomics,又称“RNA表观遗传学”)是近年来兴起的前沿科研领域。目前已知的转录后修饰已有一百多种,并且这当中许多修饰都能够参与真核生物基因表达的调控。在表观转录组学的研究当中,RNA修饰全转录组水平检测技术的发展至关重要。本论文对于真核生物mRNA上已知的几种主要修饰(包括N6-methyladenosine,N6,2’-O-dimethyladenosine, 5-methylcytidine, 5-hydroxylmethylcytidine, inosine, pseudouridine andN1-methyladenosine)的检测技术进行了综述,并且对目前表观转录组分析技术存在的挑战及未来的发展方向进行了展望。
本期 《Nature Methods》 杂志将“表观转录组分析” (Epitranscriptome analysis)评选为2016年的年度方法;伊成器研究组在该领域发表的两项化学生物学新技术(Nat. Chem. Biol. 11, 592–597 (2015) ;Nat. Chem. Biol. 12, 311–316 (2016))也被年度方法所提及。
william hill中文网博士生李笑雨(PTN-BBS联合培养项目)、北大-清华生命科学联合中心博士生熊旭深参与了论文的写作;威廉希尔伊成器研究员是该论文的通讯作者。该文章得到了国家自然科学基金、科技部973计划和北大清华生命科学联合中心的资助。
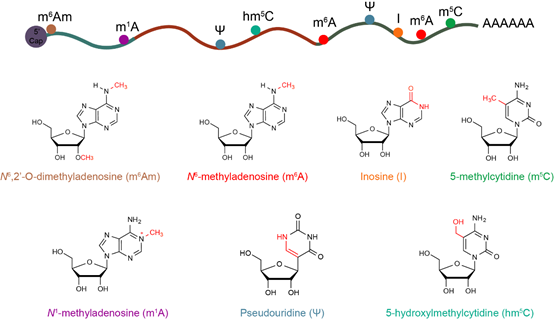
mRNA上的化学修饰 (图源于Nature Methods)
原文链接:http://www.nature.com/nmeth/journal/v14/n1/full/nmeth.4110.html