检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
CLE小肽家族是植物多肽激素中最大的一个家族,包含32个成员,目前已经报导的CLE小肽的功能多与干细胞相关,其中非常重要的一个小肽就是导管分子分化抑制因子TDIF/CLE44/CLE41,其受体为富亮氨酸重复类受体激酶PXY,TDIF-PXY信号通路控制着植物维管束中形成层细胞的复制。虽然人们对于TDIF-PXY信号通路的生物学功能已经有了一定的了解,但是有一些关键的科学问题一直没有得到解决,即TDIF小肽和受体PXY之间是如何识别的?PXY受体又是如何被TDIF小肽激活的?研究TDIF-PXY的识别以及激活的分子机理对于我们进一步理解整个CLE家族的小肽与他们的受体之间的识别和激活都具有指导意义。
威廉希尔瞿礼嘉课题组与清华大学柴继杰课题组合作,首先解析了TDIF及其受体PXY/TDR复合物晶体结构。与之前报道的线性肽采取的完全伸展的构象不一样的是,TDIF结合受体时处于一种“Ω”构象 (图一A)。保守的PxGPNP基序在这个“Ω”构象中起到关键作用。他们通过对受体PXY参与识别的氨基酸在整个CLE家族受体中进行序列比对,发现了一些在整个CLE家族受体中都非常保守的氨基酸和基序。然后他们通过体外生化实验 (图一B)、转基因植物遗传互补 (图一C) 以及结构模拟等实验,证明这些保守的氨基酸和基序确实是对于识别配体及激活信号通路至关重要。据此,他们提出了一种CLE家族受体共有的、保守的识别模式,这一识别模式对于寻找新的CLE家族小肽的受体提供了结构生物学基础和新的研究思路。
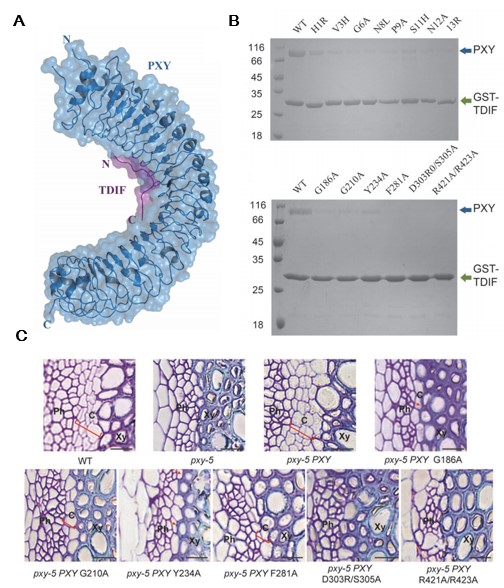
图一:
A: PXY LRR与TDIF结构图。
B: PXY LRR与TDIF关键的点突变影响两者的相互作用。
C: PXY genomic关键的点突变互补pxy-5突变体效果。
解决了识别机理之后,两个课题组又进一步探究“PXY受体是如何被TDIF小肽激活的”这一科学问题。他们合作鉴定出了TDIF-PXY信号通路的新成员SERK蛋白。SERK蛋白也是富亮氨酸重复类受体激酶,SERK家族在拟南芥中有5个成员。他们通过一系列体外以及体内相互作用实验证明,TDIF小肽可以诱导受体PXY与SERK家族蛋白相互作用(图二 A),这说明SERK蛋白可能充当的是PXY受体的共受体角色。进一步的遗传学实验证明,拟南芥serk1-1 serk2-1 bak1-5三突变体的维管束发育表型与pxy-5突变体的表型很类似,而且serk1-1 serk2-1 bak1-5对于外源施加的TDIF小肽敏感性降低(图二 B),这说明SERK家族蛋白对于PXY-TDIF信号途径介导的维管束发育是必需的。随后,他们又解析了PXY-TDIF-SERK2的晶体结构(图二 C),结构表明,TDIF小肽充当“分子胶水”(molecular glue)的功能,介导受体PXY与共受体SERKs之间的相互作用,从而激活信号途径调控拟南芥的维管束发育。至此,拟南芥中调控维管束发育的重要信号通路——PXY-TDIF-SERKs信号通路——的识别和激活机理就基本上得到了阐明。
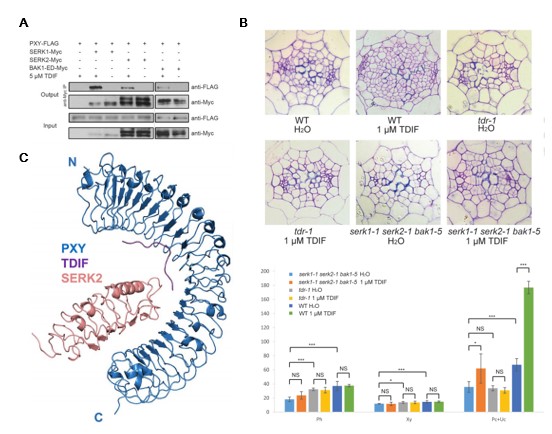
图二:
A: 在植物体内,SERK家族成员在有TDIF时与PXY相互作用。
B: serk1-1 serk2-1 bak1-5对于外源施加TDIF敏感性降低
C: PXY LRR-TDIF-SERK2 LRR三元复合体结构图。
前一项合作研究结果于2016年5月以“Crystal structure of PXY-TDIF complex reveals a conserved recognition mechanism among CLE peptide-receptor pairs”为题在线发表在国际知名学术期刊《细胞研究》(Cell Research)上,而后一项合作研究结果于2016年7月以“SERK family receptor-like kinases function as a co-receptor with PXY for plant vascular development”为题,在线发表在Cell旗下期刊《分子植物》(Molecular Plant)上。清华大学威廉希尔张贺桥博士和william hill中文网博士生林晓雅是这两篇文章的共同第一作者,英国威廉希尔公司瞿礼嘉教授和清华大学柴继杰教授是这两篇文章的共同通讯作者。该研究得到了国家自然科学基金、生物膜与膜生物工程国家重点实验室、蛋白质与植物基因研究国家重点实验室和北大-清华生命科学联合中心的资助。
原文链接:
http://www.nature.com/cr/journal/v26/n5/full/cr201645a.html
http://www.cell.com/molecular-plant/fulltext/S1674-2052(16)30136-8